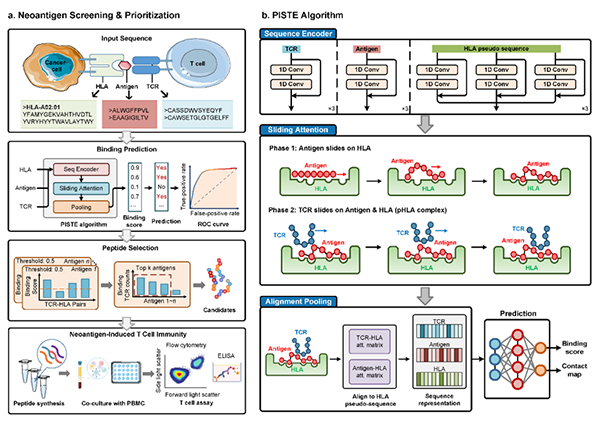
图 PISTE用于TCR-抗原- HLA结合预测和个性化新抗原筛选
在国家自然科学基金项目(批准号:82425104、81825020)等资助下,华东师范大学/华东理工大学李洪林教授团队与华东师范大学计算机学院张凯教授团队、复旦大学类脑智能科学与技术研究院张捷研究员团队、上海市第六人民医院胡晓勇主任医师团队等多个团队合作,在基于人工智能(AI)识别肿瘤新抗原方面取得进展。研究成果以“一种用于预测TCR-抗原-HLA结合的Sliding-Transformer神经构架(Sliding-attention transformer neural architecture for predicting T cell receptor-antigen-human leucocyte antigen binding)”为题,于2024年9月27日在线发表于《自然•机器智能》(Nature Machine Intelligence)杂志。论文链接:https://www.nature.com/articles/s42256-024-00901-y。
肿瘤新抗原研究的进展加速了肿瘤免疫疗法的开发,包括癌症疫苗、过继细胞疗法和抗体疗法。新抗原由肿瘤细胞基因突变产生,能激活T细胞反应,具有强免疫原性和肿瘤异质性。新一代测序和人工智能技术的进步使得快速识别和预测肿瘤新抗原成为可能。然而,识别引发强大T细胞免疫反应的新抗原仍然像大海捞针:其涉及三种高度多态性生物序列T细胞抗原受体(TCR)/抗原/人类白细胞抗原(HLA)的复杂相互作用,需要从大量序列组合中筛选出真正结合的TCR-抗原-HLA三元组;其次,构建物理上可解释且数据高效的预测方法对于增强对人类免疫反应的理解至关重要;此外,生物序列在个体中差异大,模型如何捕获残基相互作用内在模式并泛化到罕见的序列是一个重要问题。目前,仍缺乏全面的解决方案。
该研究通过构建“物理启发的Sliding-Transformer”算法(PISTE),实现TCR-抗原-HLA结合精准预测和肿瘤新抗原筛选。该算法在Transformer模型中引入了新颖的物理先验,以残基相互作用的梯度场为指引实现氨基酸残基的逐步定位,从而模拟两个(或更多个)生物序列相互滑动并形成亲和的虚拟过程,为探索TCR/抗原/HLA的复杂相互作用和人类免疫反应的分子基础提供了新的视角和方法。该算法达到了90%以上的精度,并提供残基相互作用的清晰图景。在一项前列腺癌的前瞻性研究中,75%的患者对通过PISTE预测的新抗原具有免疫反应,展现出其在临床相关的免疫应用方面的巨大潜力(图)。
该研究不仅体现了药学与人工智能、临床医学、计算科学等多学科交叉融合的优势,也展示了多学科团队合作在推动医学前沿研究中的重要作用,为人工智能赋能肿瘤新抗原疫苗自动化与智能化设计提供新思路。