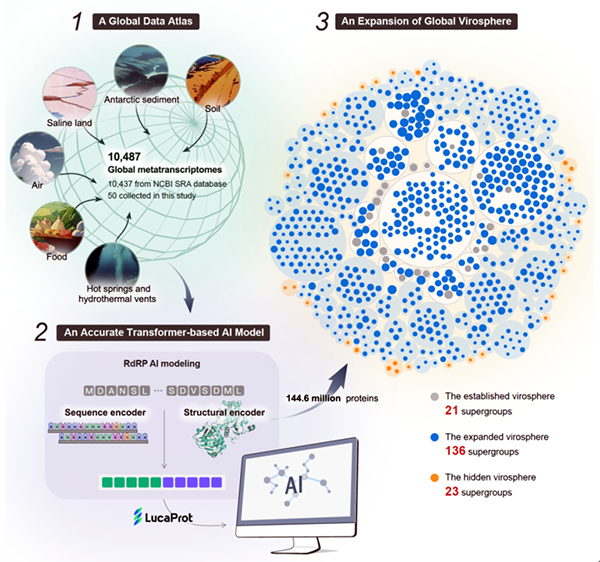
图 使用人工智能对全球病毒圈的深度挖掘
在国家自然科学基金项目(批准号:82341118、32270160)等资助下,中山大学医学院施莽教授团队、阿里云李兆融团队,联合悉尼大学Edward Holmes教授团队,在人工智能(artificial intelligence,AI)算法识别潜在的RNA病毒种(species)及RNA病毒超群(supergroups)研究方面取得进展。相关成果以“利用人工智能揭示隐藏的RNA病毒圈(Using Artificial Intelligence to Document the Hidden RNA Virosphere)”为题,于2024年10月9日在《细胞》(Cell)杂志在线发表。论文链接:https://www.cell.com/cell/fulltext/S0092-8674(24)01085-7。
病毒是地球生态系统的重要组成部分,与人类健康关系密切。目前人类对病毒多样性的认识仍然非常有限,自然界病毒圈仍有大量病毒种类处于未知状态。传统的RNA病毒鉴定方法高度依赖于序列同源性比对,即通过比较未知病毒与已知病毒的序列相似性来识别。然而,由于RNA病毒种类繁多且高度分化,这种方法难以捕捉缺乏同源性或同源性极低的病毒序列。如何高效、准确地发现和鉴定新病毒仍充满挑战。
研究团队研发了一种利用蛋白质序列和结构信息的变换器架构(Transformer)深度学习语言模型“LucaProt”,该模型具有较高的准确性(假阳性率为0.014%)和特异性(假阴性率为1.72%)。进一步,利用“LucaProt”深度挖掘来自全球各类生物环境的10,487份宏转录组数据,发现超过51万条病毒基因组,代表超过16万个潜在的RNA病毒物种和180个RNA病毒超群,使现有RNA病毒超群数量增加了约9倍(图)。其中,23个超群无法通过传统的同源性分析识别,被称为病毒圈的“暗物质”。这些新发现的病毒来自地球上的各类生态环境,包含南极底泥、深海热泉、活性污泥和盐碱滩等极端环境,显著拓宽了人们对RNA病毒分布和适宜生境的理解。此外,该研究还发现迄今为止最长的RNA病毒基因组,同时揭示了多种复杂的基因组结构,展示了RNA病毒在基因组进化上的高度灵活性。
这项研究将人工智能与病毒基因组发现结合,突破了传统的病毒分离和同源性分析方法的局限,扩展了人们对病毒圈的认知。